Cristina Sotomayor Vivas ([email protected])
Luz Rosario Guevara Cruz ([email protected])
Utilizar la base de datos KEGG (Kyoto Encyclopedia of Genes and Genomes) para identificar qué genes están involucrados más frecuentemente en la carcinogénesis viral y en qué vías de señalización se encuentran.
- Utilizamos el módulo Bio.KEGG para acceder a la vía de señalización de interés. Por medio de la función
kegg_getdel módulo Bio.KEGG.REST, accedemos a la vía por su KEGG ID. - Posteriormente, obtenemos los genes de la vía, y utilizando una versión modificada de la clase
Recordde Bio.KEGG.Gene, obtenemos la información de cada gen. Modificamos la clase para, además de la información que ya se obtiene, almacenar las vías de señalización en las que participa cada uno de esos genes. - Utilizamos la librería
pandaspara almacenar los datos obtenidos. Almacenamos los genes en unDataFrame, guardando por columnas la información acerca del código de acceso, el nombre, la posición, y las vías de señalización, los cuales se obtienen como atributos del objetoGene. - Utilizamos diversas funciones de la librería de
pandaspara analizar la información obtenida, específicamente
- Evaluamos qué genes eran los que se repetían más.
- Evaluamos qué vías de señalización asociadas a cáncer se repiten más.
- Utilizamos las librerías
seabornymatplotlibpara obtener y guardar las gráficas que representan los resultados obtenidos.
Observamos que el gen que codifica a la proteína p53 es el que más repeticiones presenta, con un total de 9.
La proteína p53 o proteína tumoral p53 es una proteína nuclear que se encarga de controlar la proliferación celular, p53 induce tanto la parada del ciclo celular que previene la replicación de DNA dañado y la apoptósis. Está clasificado como un gen supresor de tumores. Se ha encontrado que las mutaciones generadas en un alelo del gen p53 en las células somáticas se relaciona con la aparición de cáncer, sin embargo cuando la mutación se presenta en células de la línea germinal se desarrolla el síndrome Li-Fraumeni.
El gen p53 es codificado a bajos niveles en las células de manera normal, sin embargo bajo situaciones de estrés celular o daño al DNA p53, se genera un proceso de activación y modificación post-traduccional de los residuos proteicos.
 Estos resultados señalan que las mutaciones relacionadas al gen p53 son un indicio consistente para indicar la presencia o alta probabilidad para desarrollar cáncer, pues su presencia es doble al resto de mutaciones en demás genes.
Estos resultados señalan que las mutaciones relacionadas al gen p53 son un indicio consistente para indicar la presencia o alta probabilidad para desarrollar cáncer, pues su presencia es doble al resto de mutaciones en demás genes.
Los siguientes 6 genes todos corresponden a subunidades de kinasas de la familia PI3K, las cuales participan en la transducción de señales y también se han asociado a cáncer.
La vía de señalización más asociada a cáncer es FOXO, con un total de 9 repeticiones.
FOXO box es una subfamilia de factores de transcripción asociados a la mitocrondría, estos son responsables de la expresión de ligandos a receptores para la apoptósis celular. Asímismo también se encarga de regular los procesos de proliferación celular, el metabolismo de la glucosa y resistencia a estrés oxidativo. La vía de señalización encontrada en distintos tipos de cáncer que más interactúa con FOXO es la vía de señalización PIK3/AKT.
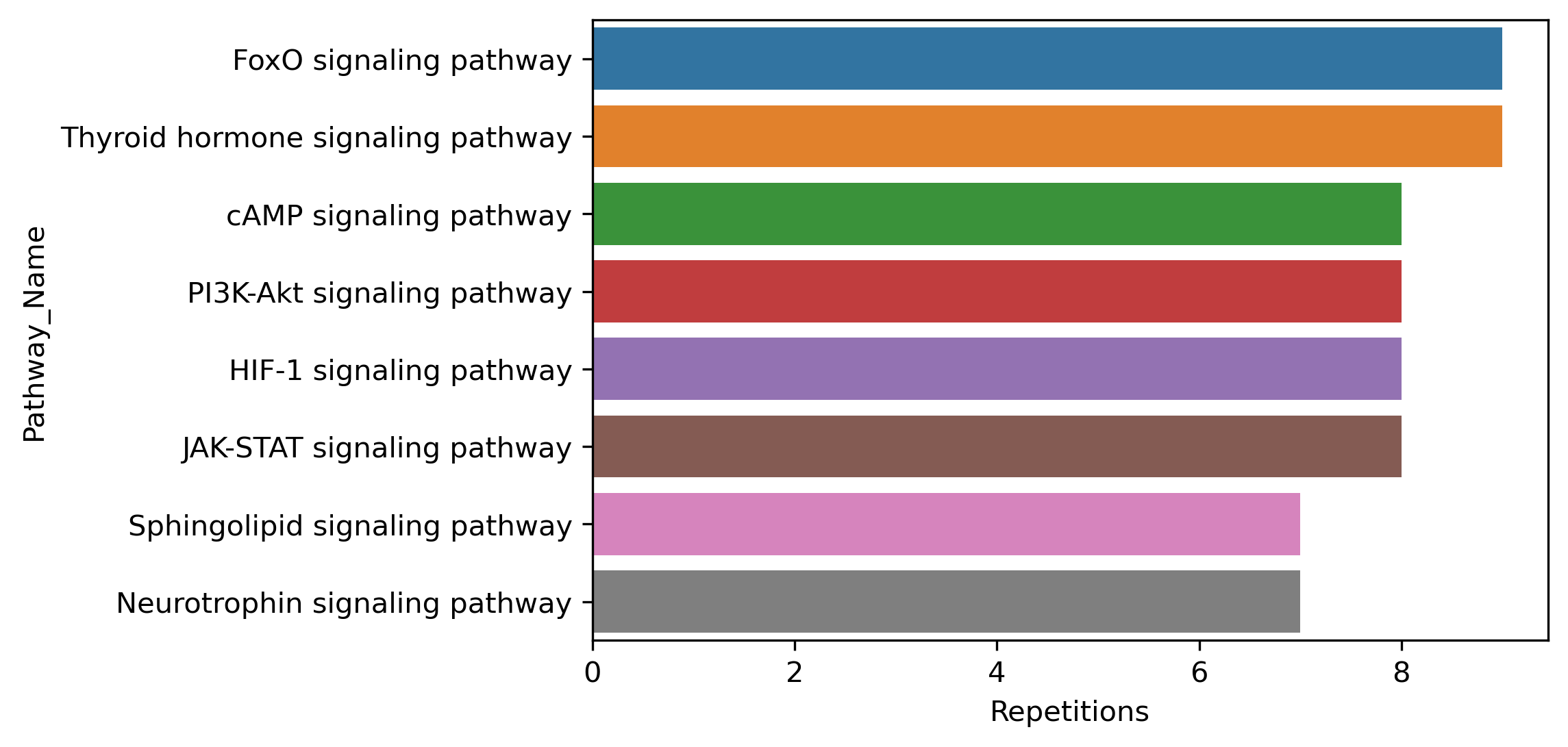 En esta gráfica podemos observar que no hay una sola vía de señalización que sea determinante para el desarrollo de cáncer, ya que una diversidad de vías de señalización se pueden encontrar involucradas en su desarrollo, y se sabe que los genes encontrados previamente pueden participar en varias vías. Resaltamos la presencia de la vía PI3K-Akt, la cual podemos esperar dados los diferentes genes relacionados a ella que encontramos en el paso previo.
En esta gráfica podemos observar que no hay una sola vía de señalización que sea determinante para el desarrollo de cáncer, ya que una diversidad de vías de señalización se pueden encontrar involucradas en su desarrollo, y se sabe que los genes encontrados previamente pueden participar en varias vías. Resaltamos la presencia de la vía PI3K-Akt, la cual podemos esperar dados los diferentes genes relacionados a ella que encontramos en el paso previo.
También analizamos las vías de señalización más repetidas al considerar todos los genes, no solo los más frecuentes, donde obtuvimos la siguiente gráfica:
 Aquí podemos observar que la frecuencia de vías de señalización cambia, mostrando a la vía fosfatidilinositol 3 kinasa (PI3K/Akt) como la que más recurrencia tiene. PI3K/ Akt está asociada a funciones como el crecimiento celular y la inhibición de la apoptosis. El funcionamiento de esta vía está regulado por la estimulación de más factores moleculares y proteicos que son resultado del crecimiento celular; la mutación o cambios en la expresión de estos factores reguladores promueven la activación permantente en la vía PI3K, favoreciendo una actividad de metástasis tumoral y resistencia a tratamientos anticancerígenos.
Aquí podemos observar que la frecuencia de vías de señalización cambia, mostrando a la vía fosfatidilinositol 3 kinasa (PI3K/Akt) como la que más recurrencia tiene. PI3K/ Akt está asociada a funciones como el crecimiento celular y la inhibición de la apoptosis. El funcionamiento de esta vía está regulado por la estimulación de más factores moleculares y proteicos que son resultado del crecimiento celular; la mutación o cambios en la expresión de estos factores reguladores promueven la activación permantente en la vía PI3K, favoreciendo una actividad de metástasis tumoral y resistencia a tratamientos anticancerígenos.
También obtuvimos una gráfica para representar los genes asociados a estas vías de señalización comprometidas con más ocurrencias.
Al analizar la ocurrencia de los genes con más repeticiones en estas vías, se puede resaltar la presencia de fosfatidilinositol-4,5-bifosfato, un componente de la membrana fosfolipídica que se asocia al funcionamiento de la señalización celular así como la modulación de canales iónicos asociados a la liberación de neurotransmisores y funciones fisiológicas. La interacción entre fosfatidilinositol-4,5-bifosfato y los canales iónicos permite mantener una corriente constante entre el cambio de gradientes de los canales iónicos. En la gráfica observamos que las subunidades moleculares alpha, beta y delta tienen el mismo número de apariciones asociadas a carcinogénesis cuando nos enfocamos en estas vías de señalización.
Las mutaciones asociadas al gen p53 y la vía de señalización FOXO tienen evidencia cuantitativa de ser variaciones asociadas a la supresión de tumores, y los residuos proteicos alterados son comúnmente aislados de tumores. Por medio de este proyecto, vimos cómo podemos recopilar diferentes tipos de información de la base de datos KEGG, en este caso genes y vías metabólicas, para obtener información de los mecanismos involucrados en esta enfermedad. Podríamos repetir este proceso para diferentes vías metabólicas, solo cambiando el código de acceso de cada vía, e incluso modificar el código para recopilar más información, por ejemplo, incluir los links que da KEGG a otras bases de datos, y usar otros módulos como Bio.Entrez para obtener información de otras bases. El saber cómo trabajar con diferentes bases de datos a través de programas que nosotros mismos creamos es una herramienta muy útil y muy versátil para acceder y manejar información, y el uso de herramientas como pandas, matplotlib y seaborn amplía lo que podemos llevar a cabo con esos datos.
- Farhan, M., Wang, H., Gaur, U., Little, P., Xu J., Zheng, W. (2017) FOXO Signaling Pathways as Therapeutic Targets in Cancer, NCBI. doi: 10.7150/ijbs.20052
- Silva, A., Gutiérrez, A., Arias, C., Lazaro, I. (2006) Estructura, regulación y funciones del gen supresor de tumores p53, Congreso virtual Hispanoamérica de Anatomía Patológica. https://conganat.uninet.edu/conferencias/C016/index.html
- Carter, M., Brunet, A. (2007) FOXO transcription factors, NCBI. doi:10.1016/j.cub.2007.01.008
- Pinzón, Carlos Eduardo, Serrano, Martha Lucía, & Sanabria, María Carolina. (2009). Papel de la vía fosfatidilinositol 3 kinasa (PI3K/Akt) en humanos. Revista Ciencias de la Salud, 7(2), 47-66. Retrieved December 02, 2021, from http://www.scielo.org.co/scielo.php?script=sci_arttext&pid=S1692-72732009000200007&lng=en&tlng=es.
- Pérez, A., Alamilla, J.(2010) El fosfatidilinositol-4,5-bifosfato y sus acciones sobre los canales iónicos. Revista Biomed. https://www.medigraphic.com/pdfs/revbio/bio-2010/bio102f.pdf

